
Produire de nouveaux
savoirs
Explorer de nouvelles avenues pour développer la connaissance médicale de demain dans une approche qui intègre recherche fondamentale et recherche clinique.
Nos groupes de recherche sont dirigés par des chercheuses et chercheurs principaux qui collaborent dans un esprit de collégialité et de partage pour rapprocher la recherche des patients et patientes et former la relève scientifique. Ce sont des esprits libres et indépendants qui travaillent sans relâche pour améliorer la santé. Découvrez les chercheurs et chercheuses de l'IRCM.
Recherche en cours
« Notre laboratoire s’intéresse à la caractérisation des interactions protéine-protéine, aux mécanismes d'assemblage des complexes protéiques et de leur modulation dans les cellules malades »
Benoit Coulombe, directeur de recherche
Protéomique translationnelle
BIO - Benoit Coulombe est directeur de l'unité de recherche en protéomique translationnelle à l'Institut de Recherches Cliniques de Montréal (IRCM), où il occupe également le poste de responsable de la Conduite Responsable en Recherche (CRC). Coulombe a obtenu un doctorat en biologie moléculaire de l'Université de Montréal et a effectué des recherches postdoctorales à l'Université de Toronto avec Jack Greenblatt (biochimie) et à l'Université libre de Bruxelles, en Belgique, avec Albert Goldbeter (chimie). Depuis 2022, il est rédacteur-en-chef de section pour la revue en libre accès Molecular Brain (Springer Nature). Coulombe a publié plus de 100 articles de recherche et ses travaux ont été cités plus de 8 300 fois. Il est également membre du groupe de travail (Taskforce) sur l'enregistrement des essais cliniques et la communication de leurs résultats, qui préconise le libre accès aux résultats de tous les essais cliniques passés et présents. Ses recherches portent sur la caractérisation des interactions protéine-protéine, les mécanismes d'assemblage des complexes protéiques et leur modulation dans les cellules malades
| Équipe |
|
| Affiliations |
|
| Formations et expérience |
|

PAIRS est un algorithme qui utilise le système d'IA AlphaFold-Multimer (DeepMind/Google) pour identifier les interactions physiques directes protéine-protéine d'origine humaine ou virale. Précédemment appelé AlphaFold-pairs, PAIRS a été développé par le laboratoire de protéomique translationnelle de l'Institut de recherches cliniques de Montréal (IRCM).

Article le plus récent
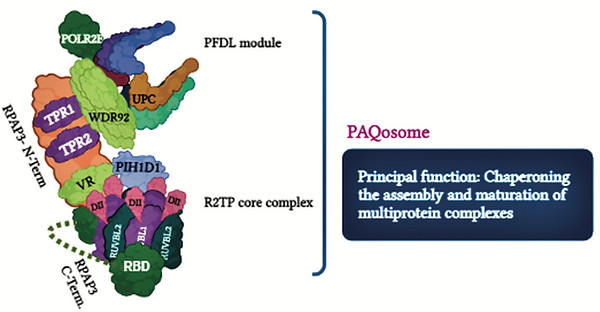
Several drugs target the PAQosome subunits
Pinard M, Moursli A, Coulombe B.
Drugs targeting the particle for arrangement of quaternary structure (PAQosome) and protein complex assembly
Expert Opin Drug Discov. 2023 Oct 15:1-15. doi: 10.1080/17460441.2023.2267974
Articles vedettes
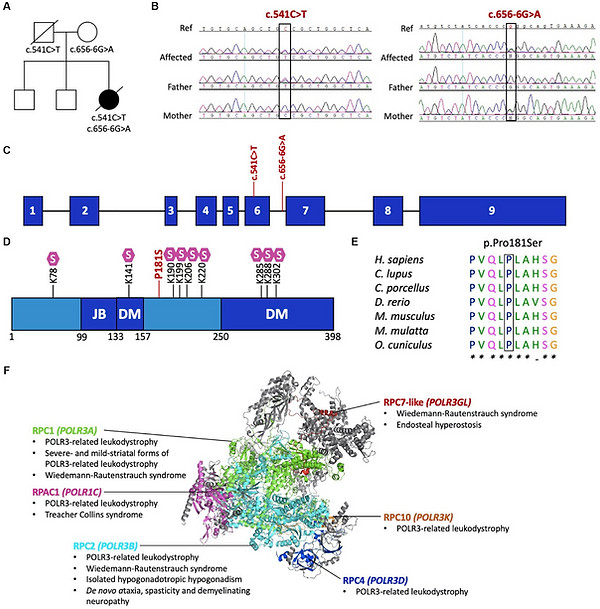
Variants of RNA polymerase III subunits strike again and cause leukodystrophy
Macintosh J, Perrier S, Pinard M, Tran LT, Guerrero K, Prasad C, Prasad AN, Pastinen T, Thiffault I, Coulombe B, Bernard G.
Biallelic pathogenic variants in POLR3D alter tRNA transcription and cause a hypomyelinating leukodystrophy: A case report
Front Neurol. 2023 Oct 13. doi: 10.3389/fneur.2023.1254140
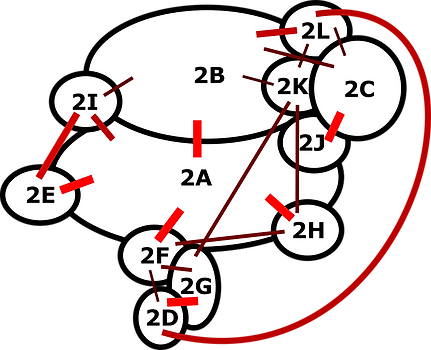
AI predicts protein direct physical interactions with AlphaFold-pairs
Poitras C, Lamontage F, Grandvaux N, Song H, Pinard M, Coulombe B.
High-accuracy mapping of human and viral direct physical protein-protein interactions using the novel computational system AlphaFold-pairs
bioRxiv. 2023 Aug 30:2023.08.29.555151. doi: 10.1101/2023.08.29.555151. Preprint.
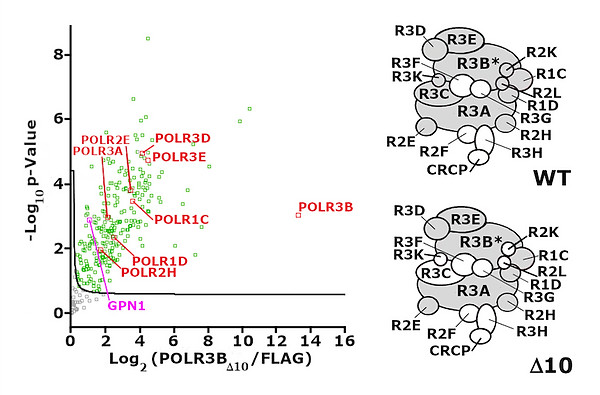 A mouse model to study RNA polymerase III-related leukodystrophies
A mouse model to study RNA polymerase III-related leukodystrophies
Michell-Robinson M, Watt KEN, Grouza V, Macintosh J, Pinard M, Tuznik M, Chen X, Darbelli L, Wu CL, Perrier S, Chitsaz D, Uccelli NA, Liu H, Cox TC, Müller C, Kennedy TE, Coulombe B, Rudko D, Trainor PA, Bernard G.
Hypomyelination, hypodontia and craniofacial abnormalities in a Polr3b mouse model of leukodystrophy
Brain. 2023 Aug 28:awad249. doi: 10.1093/brain/awad249
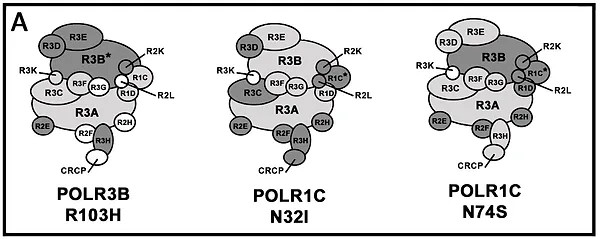 Discovery of a drug that stimulates protein complex assembly
Discovery of a drug that stimulates protein complex assembly
Pinard M, Dastpeyman S, Poitras C, Bernard G, Gauthier MS, Coulombe B.
Riluzole partially restores RNA polymerase III complex assembly in cells expressing the leukodystrophy-causative variant POLR3B R103H
Mol Brain. 2022 Nov 30;15(1):98. doi: 10.1186/s13041-022-00974-z.
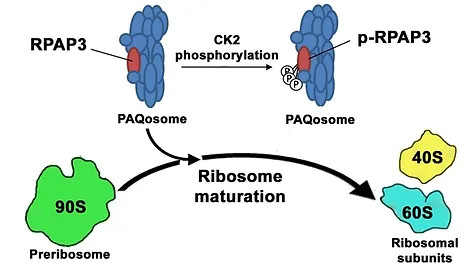
Discovery of a novel mode of substrate selection by the PAQosome
Pinard M, Cloutier P, Poitras C, Gauthier MS, Coulombe B.
Unphosphorylated Form of the PAQosome Core Subunit RPAP3 Binds Ribosomal Preassembly Complexes to Modulate Ribosome Biogenesis
J Proteome Res. 2022 Apr 1;21(4):1073-1082. doi: 10.1021/acs.jproteome.1c00938
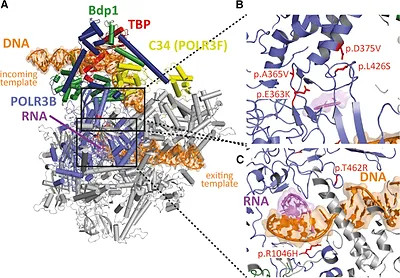
Discovery of a novel neurodegenerative disorder and causative mutations
Djordjevic D, Pinard M, Gauthier MS, Smith-Hicks C, Hoffman TL, Wolf NI, Oegema R, van Binsbergen E, Baskin B, Bernard G, Fribourg S, Coulombe B, Yoon G.
De novo variants in POLR3B cause ataxia, spasticity, and demyelinating neuropathy
Am J Hum Genet. 2021 Jan 7;108(1):186-193. doi: 10.1016/j.ajhg.2020.12.002
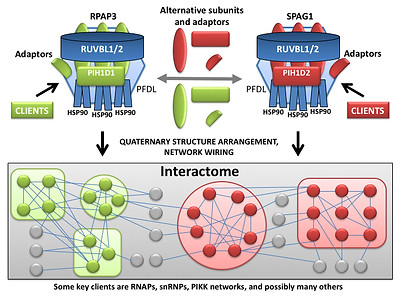 A mechanistic model for interactome biogenesis
A mechanistic model for interactome biogenesis
Coulombe B, Cloutier P, Gauthier MS.
How do our cells build their protein interactome?
Nat Commun. 2018 Jul 27;9(1):2955. doi: 10.1038/s41467-018-05448-2.
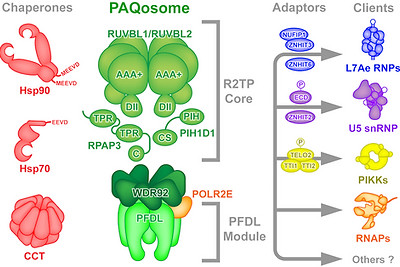
Summary of the PAQosome structure and function
Houry WA, Bertrand E, Coulombe B.
The PAQosome, an R2TP-Based Chaperone for Quaternary Structure Formation
Trends Biochem Sci. 2018 Jan;43(1):4-9. doi: 10.1016/j.tibs.2017.11.001.
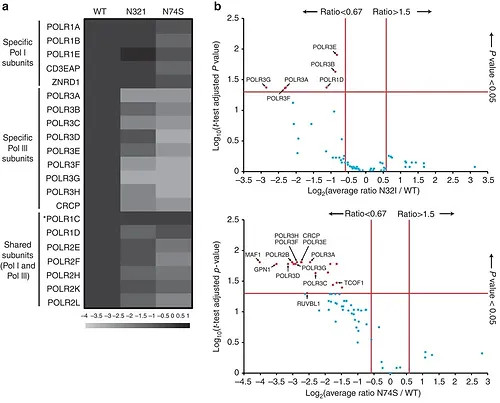
Discovery of leukodystrophy-causing mutations that target assembly of RNA pol III
Thiffault I, Wolf NI, Forget D, Guerrero K, Tran LT, Choquet K, Lavallée-Adam M, Poitras C, Brais B, Yoon G, Sztriha L, Webster RI, Timmann D, van de Warrenburg BP, Seeger J, Zimmermann A, Máté A, Goizet C, Fung E, van der Knaap MS, Fribourg S, Vanderver A, Simons C, Taft RJ, Yates JR 3rd, Coulombe B, Bernard G.
Recessive mutations in POLR1C cause a leukodystrophy by impairing biogenesis of RNA polymerase III
Nat Commun. 2015 Jul 7;6:7623. doi: 10.1038/ncomms8623.
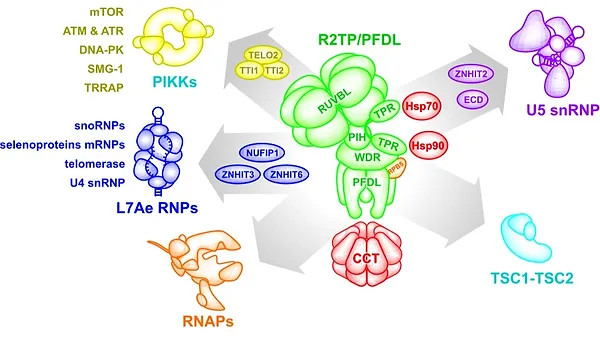 Discovery of molecular adaptors for substrate selection by the PAQosome
Discovery of molecular adaptors for substrate selection by the PAQosome
Cloutier P, Poitras C, Durand M, Hekmat O, Fiola-Masson É, Bouchard A, Faubert D, Chabot B, Coulombe B.
R2TP/Prefoldin-like component RUVBL1/RUVBL2 directly interacts with ZNHIT2 to regulate assembly of U5 small nuclear ribonucleoprotein
Nat Commun. 2017 May 31;8:15615. doi: 10.1038/ncomms15615.
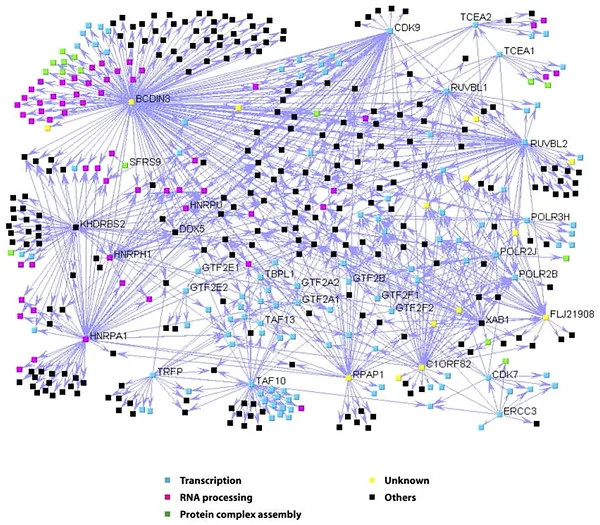 Using machine learning to build protein interaction networks
Using machine learning to build protein interaction networks
Jeronimo C, Forget D, Bouchard A, Li Q, Chua G, Poitras C, Thérien C, Bergeron D, Bourassa S, Greenblatt J, Chabot B, Poirier GG, Hughes TR, Blanchette M, Price DH, Coulombe B.
Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzyme
Mol Cell. 2007 Jul 20;27(2):262-274. doi: 10.1016/j.molcel.2007.06.027.
Découvertes importantes
Outils
| Revue de presse |
|
| Revue de presse |
|
| Subventions |
|
| Reconnaissances et honneur |
Rejoindre
le groupe
de recherche
Postes disponibles
Études cliniques en cours
Partenaires de recherche
Contacter le
groupe de recherche
514 987-5662
benoit.coulombe@ircm.qc.ca
La vie au
laboratoire

© Institut de recherches cliniques de Montréal, Année.Tous droits réservés. | Politique sur la protection des renseignements personnels | Conditions d'utilisation | Réalisation Agence Riposte


